
Research
〜時空間的な遺伝子発現のしくみを解き明かす〜
オキラボでは、個体発生や細胞分化、疾患などにおける時空間的な遺伝子発現のしくみを解き明かすことを目標とし、そのための実験技術とデータ基盤を開発しています。我々はPICという空間トランスクリプトーム技術を開発し、局所的に発現する遺伝子を網羅的に解析します。また、ChIP-Atlasというエピゲノミクス統合データベースを開発し、データ駆動型アプローチによって遺伝子発現の制御機構を解き明かします。また両者を融合することで、時空間的な遺伝子発現の制御機構という観点から細胞分化、疾患や薬効のメカニズムを解明します。
Theme 1:PICによる空間トランスクリプトーム解析
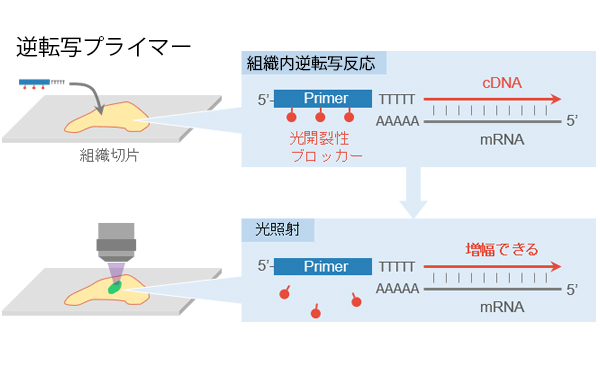

私たちはPIC(Photo-Isolation Chemistry)という技術を開発し、微小な細胞集団や領域における遺伝子発現情報を世界最高レベルの解像度と検出感度でピックアップすることに成功しました(Honda, et al. Nat Commun. 2021)。これは、目的の細胞集団や微小組織に光を照射すると、そこで発現する遺伝子を網羅的に検出できる、画期的な技術です。これにより、脳のさまざまな領域に光を照射して、各領野で特異的に発現する遺伝子を検出することに成功しています。さらに、マウス胎児の非常に小さな細胞集団や、核内のミクロン以下の微小構造体からも遺伝子の網羅的な検出に成功しています。現在PICをさらに発展させ、微小領域の遺伝子発現だけでなく、エピゲノム情報をピックアップするための技術開発を進めています。
Theme 2:ChIP-Atlasによる遺伝子制御機構の解明
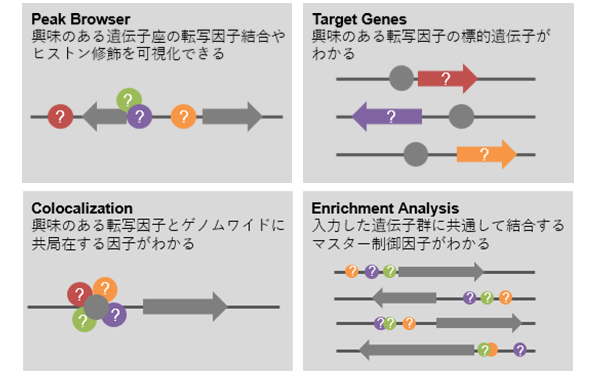
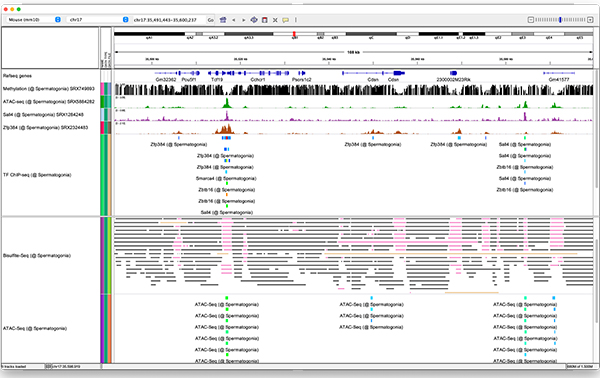
私たちは、世界中から報告されたChIP-seqデータを全て統合したデータベース、ChIP-Atlasを開発しています(Oki, et al. EMBO Rep. 2018)。修飾ヒストンや転写因子など、約14万件ものChIP-seq実験データを統合解析し、のべ28億件のタンパク質-ゲノム相互作用情報を抽出しています。この膨大なデータはChIP-AtlasのWebサイトを通じて公開していますが、誰でも簡単に閲覧や解析ができるため、世界中の研究者に使われ、多くの論文に引用されています。今後、オープンクロマチン情報やゲノムメチル化情報も収集することにより、時空間的な遺伝子発現を司るエピジェネティックな制御機構を解明します。
Theme 3:新技術とデータ解析を融合した応用研究
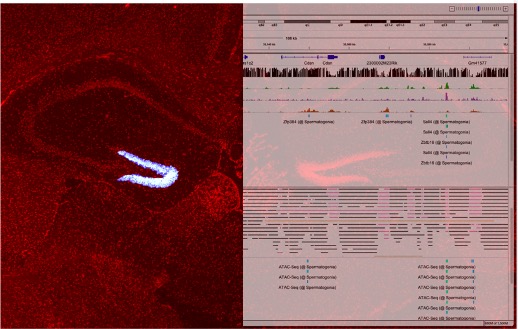
PICはあらゆる臓器や組織において、細胞タイプ特異的な遺伝子発現を抽出することができます。さらに、その遺伝子群をChIP-Atlasでデータ解析すれば、それらを統合的に制御する転写因子を特定することができます。これにより、各種細胞タイプの分化を司るマスター制御因子が特定されるため、ダイレクトプログラミングや再生医療への応用が期待されます。
また、投薬によって発現が変動する遺伝子群をChIP-Atlasで解析し、薬剤で変動する遺伝子のマスター制御因子の特定を進めています。つまり薬剤の作用機序のコアとなる転写因子を理解できるため、薬剤の標的因子の推定や新たな薬効の予測に応用できます。さらに遺伝性疾患の原因となるSNPに、どんな転写因子が結合するかをChIP-Atlasで探索しています。これにより発症プロセスに関わる転写因子を推定できるため、効果的な薬剤候補の選定に応用できます。